
von Dr. Wollschläger, Institut für Klinikhygiene, Medizinische Mikrobiologie und Klinische Infektiologie Klinikum Nürnberg)
Durch diese spezielle PCR können Einzelnukleotid-Polymorphismen (Single-nucleotide polymorphism, SNP) in der genomischen Sequenz von SARS-COV2 differenziert werden. Hierfür werden Sonden verwendet, die den Farbstoff Fluorescein (FAM) enthalten, welche eine erhöhte Fluoreszenz aufweisen, sobald dieser an die cDNA bindet. Weil die Sonden bereits spezifisch für die entsprechende Mutation ausgelegt sind (z.B. N501Y), erhält man meistens nur im Falle einer vorhandenen Mutation eine detektierbare Amplifikation.
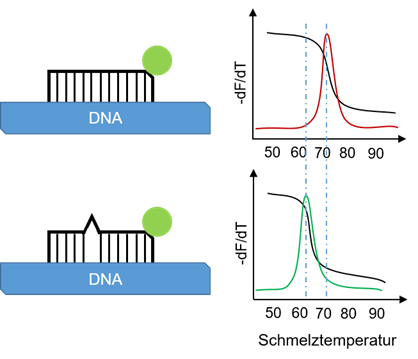
Da der Test ein sondenbasierter Assay ist, kann man dank der variantenspezifischen Sonden durch eine anschließende Schmelzkurvenanalyse Rückschlüsse auf den vorhandenen SNP ziehen.
Hierfür wird die Fluoreszenz gemessen während die Probe langsam aufgeheizt wird. Sobald die Sonden sich lösen, ist eine Abnahme der Fluoreszenz messbar (Abbildung, schwarze Kurve). In der ersten negativen Ableitung ergibt sich an der steilsten Stelle ein Peak (Abbildung, rote Kurve).
Binden die Sonden auf Grund von Fehlpaarungen nicht korrekt, ist die Bindung zur cDNA schwächer und der Schmelzpunkt folglich niedriger (Abbildung, grüne Kurve). Prinzipiell ist dieser Test für fast jeden SNP anwendbar, jedoch werden die entsprechenden spezifischen Sonden benötigt, welche ggf. über andere Schmelzpunkte verfügen.
Weitere Fragen dazu beantwortet Ihnen gern Frau Roswitha Kerl, Landesvertretung Laboratoriumsmedizin Bayern.












